Comprendre les processus évolutifs des mycobactéries pathogènes pour prévenir l’émergence de variants à danger sanitaire élevé
Crédits : A. Le Meur
Soutenance de thèse
25/11/2025
09:30:00
Adrien Le Meur, ESE
IDEEV - Salle Rosalind Franklin
Thèse dirigée par Guislaine Refrégier (ESE, équipe Génétique et Ecologie Evolutives .
Résumé
Mycobacterium tuberculosis est une bactérie pathogène responsable de la tuberculose. C’est la première cause de mortalité dans le monde par un agent infectieux. Le traitement pour la tuberculose est un régime de plusieurs antibiotiques qui doivent être pris chaque jour pendant plusieurs mois, ce qui limite son effcacité dans des pays à ressources limitées. La bactérie est capable d’acquérir de manière spontanée des résistances aux antibiotiques. Pour suivre l’évolution de la maladie et diagnostiquer la résistance aux antibiotiques, M. tuberculosis est beaucoup séquencée. Plus de 200,000 archives de séquençage sont disponibles publiquement dans des bases de données. Pendant ma thèse, j’ai développé Genotube, un pipeline d’analyse bioinformatique permettant d’identifier des variants à partir de données de séquençage Illumina, de prédire la résistance aux antibiotiques et d’identifier la lignée des souches.
A l’aide de ce pipeline, nous avons étudié plus de 50,000 archives de séquençage disponibles publiquement. Nous avons identifié des mutations dans les gènes impliqués dans la réparation, réplication et recombinaison du génome associés à la résistance aux antibiotiques. Nous avons montré que ces mutations étaient associées à un plus grand taux d’évolution moléculaire et une fréquence plus importante d’acquisition de résistances aux antibiotiques dans l’espèce proche Mycobacterium smegmatis. La présence de ces phénotypes mutateurs pourraient jouer un rôle dans l’apparition de la résistance aux antibiotiques dans M. tuberculosis.
Nous avons également utilisé Genotube sur des jeux de données plus petits. Nous avons confirmé la présence d’une nouvelle lignée de M. tuberculosis. Nous avons également analysé des échantillons provenant d’enfants atteints de la tuberculose en Ile de France. Nous avons identifié des transmissions récentes entre plusieurs enfants de même origine ou traités dans les même centres hospitaliers.
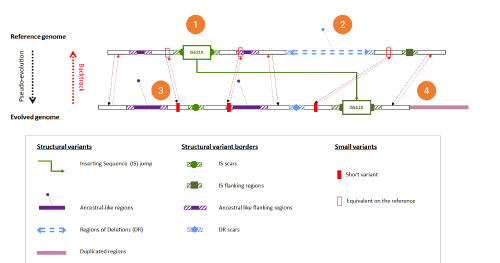
Enfin, nous avons étudié les biais et les artefacts introduits par l’analyse des données de séquençage. Les méthodes d’évaluation standards ne tiennent pas compte la diversité des variations structurales des génomes. Nous avons développé Maketube, un outil permettant de générer des génomes de test plus réalistes. Maketube introduit des grandes insertions, de grandes délétions, une grande région du pliquée et la transposition d’éléments mobiles tel que décrits pour M. tuberculosis. Nous montrons que les génomes produits par Maketube sont plus divers que les génomes produits par les méthodes standards et que cette diversité ressemble à celle des souches de M. tuberculosis. En utilisant ces génomes, nous montrons que les pipelines d’analyse bioinformatiques sont très précis mais moins sensibles. Nous montrons également que les duplications et les insertions sont une source importante de faux négatifs, mais pas les délétions. Ces résultats permettront aux chercheurs de choisir entre plusieurs méthodes bioinformatiques et écarter les variants artéfactuels sur la base de ces résultats empiriques.
Composition du jury
- Maria Laura BOSCHIROLI, directrice de recherche ANSES, rapportrice et examinatrice
- Olivier TENAILLON, directeur de recherche INSERM, rapporteur et examinateur
- Thibaut MOREL-JOURNEL, professeur associé, examinateur
- Gaelle LELANDAIS, professeure associée, examinatrice
- Guislaine REFREGIER, professeure associée, directrice de thèse