
Matthieu FALQUE
dernières mises à jour 01/01/0001
-
01 69 33 23 64
matthieu.falque@inrae.fr
Atelier Cartographie, Expression et Polymorphisme
Biologie de l'Adaptation et Systèmes en Évolution
INRAE, Ingénieur de Recherche
génétique végétale, marqueurs moléculaires, cartographie génétique, meiose et recombinaison, modélisation
publications - orcid
Parcours
- Actuellement membre de l’équipe BASE, affilié au LABEX Saclay Plant Sciences
- HDR (habilitation à diriger les recherches) 2015, Université d’Orsay
- Responsable de l’équipe ACEP (Atelier Cartographie Expression Polymorphisme)
- Depuis 1998: Ingénieur de Recherche (INRA) at UMR GQE - Le Moulon, Gif-sur-Yvette, France
- Postdoc en génétique des populations végétales (1997-1998) CNRS, Lille France
- Postdoc en génétique des populations végétales (1995-1997) NIOO-CTO, Heteren-Wageningen, Pays-Bas
- Postdoc en génétique moléculaire végétale (1994-1995) CIRAD, Montpellier, France
- Ph.D. en biologie végétale (1994), Univ Toulouse, France
Intérêts
Mes recherches dans l’équipe BASE
2 voies de formation des crossing-overs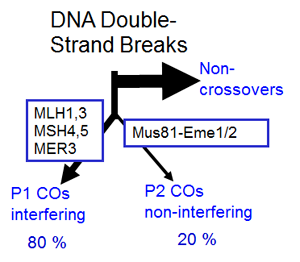
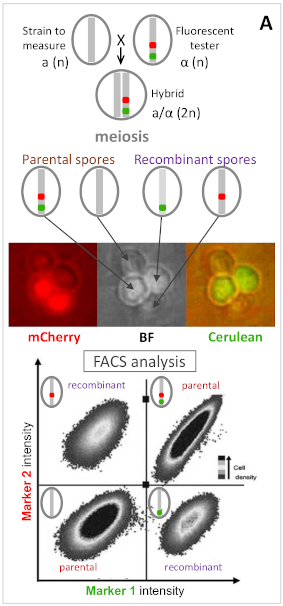
Phénotypage de la recombinaison par FACS (cas simple davec 2 marqueurs)
Plus récemment, avec Xavier Raffoux qui a effectué sa thèse avec moi, nous avons développé des approches expérimentales utilisant la levure de bière comme modèle pour étudier le déterminisme génétique de la variabilité du taux de recombinaison et de son profil le long des chromosomes.
Nojs avons d’abord développé une méthode à haut-débit pour mesurer le taux de recombinaison et l’interférence grâce à des marqueurs fluorescents. Nous avons ainsi développé 8 souches “testeurs” couvrant entièrement les chromosomes VI et XI, et partiellement le chromosome I. Dans chaque testeur, trous marqueurs fluorescents ont été intégrés dans le génome à environ 30 cM l’un de l’autre, délimitant ainsi deux intervalles adjacents à partir desquels l’interférence peut être mesurée très précisément
(Ref)
.
Nous avons ensuite appliqué cette méthode pour caractériser la diversité intra-spécifique du taux de recombinaison et de l’interférence au sein de l’espèce S. cerevisiae. Dans les différents intervalles analysés, le taux de recombinaison a montré des variations importantes entre souches, mais les profils de recombinaison le long des chromosomes varie moins. Nous avons aussi étudié le taux de recombinaison dans une région sur 10 hybrides issus de cing parents très divergents. Nos résultats indiquent globalement que le taux de recombinaison est corrélé positivement avec la similarité de séquence entre chromosomes homologues. Nous avons également estimé que les effets en cis et en trans expliquent respectivement 38% et 17% de la variance du taux de recombinaison
(Ref)
.
Mon principal intérêt de recherche maintenant concerne les aspects évolutifs de la recombinaison méiotique. En utilisant notre méthode basée sur le FACS, nous allons faire évoluer des populations de levure sous sélection récurrente pour le taux de recombinaison, et nous utiliserons ces populations évoluées pour étudier le rôle que joue la recombinaison dans la dynamique de l’adaptation au stress.
Mes recherches dans l’équipe ACEP
L’équipe transversale ACEP est une plateforme interne de service pour la construction de cartes génétiques, les analyses de polymorphisme de l’ADN et d’expression génique. Nous fournissons un service d’analyse de marqueurs moléculaires et assurons la veille technologique sur les nouvelles technologies de marquage. Je produis aussi des cartes génétiques à haute-densité sur différentes espèces de plantes, en collaboration avec d’autres laboratoires.
Dans le cadre d’ACEP, j’ai aussi développé des outils pour :
- construire des cartes génétiques (REF) et (REF)
- détecter des CNV (Copy Number Variants) à partir de données de segrégation (REF)
- mesurer à haut-débit la cinétique de croissance de micro-organismes (REF)
- calculer automatiquement des taux de recombinaison et coefficients de coincidence à partir de données de cytométrie de flux (REF) produites avec les souches fluorescentes développées précédemment (Ref)
Projets en cours
- Dans le cadre du projet EVOLREC que je coordonne (REF) :
- Evolution expérimentale sous sélection divergent récurrente pour le taux de recombinaison méiotique
- Detection et validation fonctionnelle de QTLs de recombinaison méiotique par séquençage des populations évoluées, coll. Gianni Liti (REF)
- Evolution expérimentale sous stress salin à partir de populations avec des niveaux de recombinaison variables
- Criblage d’une banque de surexpression chez S. cerevisiae pour rechercher des modulateurs de l’interférence génétique
Activités d’enseignement
Enseignement occasionnel pour des étudiants et des sélectionneurs de sociétés privées, sur le marquage moléculaire et la cartographie génétique appliqués à l’amélioration des plantes.
Publications
2024
- Falque M. , Bourgais A., Dumas F., De Carvalho M., Diblasi C.. (2024) MiniRead: A simple and inexpensive do‐it‐yourself device for multiple analyses of micro‐organism growth kinetics. Yeast, yea.3932
2022
- Hsu YM., 2022-09, Mining genetic diversity for tomorrow's agriculture, Theses, Université Paris-Saclay
- Hsu YM., Falque M. , Martin OC.. (2022) Quantitative modelling of fine-scale variations in the Arabidopsis thaliana crossover landscape. Quant Plant Bio., (3) e3
- Vidal A., Gauthier F., Rodrigez W., Guiglielmoni N., Leroux D., Chevrolier N., Jasson S., Tourrette E., Martin OC., Falque M. . (2022) SeSAM: software for automatic construction of order-robust linkage maps. BMC Bioinformatics, 1 (23) 499
2021
- Olvera-Vazquez SG., Alhmedi A., Miñarro M., Shykoff JA., Marchadier E., Rousselet A., Remoué C., Gardet R., Degrave A., Robert P., Chen X., Porchier J., Giraud T., Vander-Mijnsbrugee K., Raffoux X., Falque M. , Alins G., Didelot F., Beliën T., Dapena E., Lemarquand A., Cornille A.. (2021) Experimental test for local adaptation of the rosy apple aphid (Dysaphis plantaginea) to its host (Malus domestica) and to its climate in Europe. PCI Ecology, (Pre-registration version)
- Olvera-Vazquez SG., Remoué C., Venon A., Rousselet A., Grandcolas O., Azrine M., Momont L., Galan M., Benoit L., David GM., Alhmedi A., Beliën T., Alins G., Franck P., Haddioui A., Jacobsen SK., Andreev R., Simon S., Sigsgaard L., Guibert E., Tournant L., Gazel F., Mody K., Khachtib Y., Roman A., Ursu TM., Zakharov IA., Belcram H., Harry M., Roth M., Simon JC., Oram S., Ricard JM., Agnello A., Beers EH., Engelman J., Balti I., Salhi-Hannachi A., Zhang H., Tu H., Mottet C., Barrès B., Degrave A., Razmjou J., Giraud T., Falque M. , Dapena E., Miñarro M., Jardillier L., Deschamps P., Jousselin E., Cornille A.. (2021) Large-scale geography survey provides insights into the colonization history of a major aphid pest on its cultivated apple host in Europe, North America and North Africa. Peer Community Journal, (1) e34
- Tourrette E., Falque M. , Martin OC.. (2021) Enhancing backcross programs through increased recombination. Genet Sel Evol, 1 (53) 25
2020
- Carrillo-Perdomo E., Vidal A., Kreplak J., Duborjal H., Leveugle M., Duarte J., Desmetz C., Deulvot C., Raffiot B., Marget P., Tayeh N., Pichon JP., Falque M. , Martin OC., Burstin J., Aubert G.. (2020) Development of new genetic resources for faba bean (Vicia faba L.) breeding through the discovery of gene-based SNP markers and the construction of a high-density consensus map. Sci Rep, 1 (10) 6790
- Falque M. , Jebreen K., Paux E., Knaak C., Mezmouk S., Martin OC.. (2020) CNVmap: A Method and Software To Detect and Map Copy Number Variants from Segregation Data. Genetics, 3 (214) 561-576
2019
- Courret C., Gérard PR., Ogereau D., Falque M. , Moreau L., Montchamp-Moreau C.. (2019) X-chromosome meiotic drive in Drosophila simulans: a QTL approach reveals the complex polygenic determinism of Paris drive suppression. Heredity, 6 (122) 906-915
- Fustier MA., Martínez-Ainsworth NE., Aguirre-Liguori JA., Venon A., Corti H., Rousselet A., Dumas F., Dittberner H., Camarena MG., Grimanelli D., Ovaskainen O., Falque M. , Moreau L., Meaux J., Montes-Hernández S., Eguiarte LE., Vigouroux Y., Manicacci D., Tenaillon MI.. (2019) Common gardens in teosintes reveal the establishment of a syndrome of adaptation to altitude. PLOS Genetics, 12 (15) e1008512
- Kreplak J., Madoui MA., Cápal P., Novák P., Labadie K., Aubert G., Bayer PE., Gali KK., Syme RA., Main D., Klein A., Bérard A., Vrbová I., Fournier C., d’Agata L., Belser C., Berrabah W., Toegelová H., Milec Z., Vrána J., Lee HT., Kougbeadjo A., Térézol M., Huneau C., Turo CJ., Mohellibi N., Neumann P., Falque M. , Gallardo K., McGee R., Tar’an B., Bendahmane A., Aury JM., Batley J., Le Paslier MC., Ellis N., Warkentin TD., Coyne CJ., Salse J., Edwards D., Lichtenzveig J., Macas J., Doležel J., Wincker P., Burstin J.. (2019) A reference genome for pea provides insight into legume genome evolution. Nat Genet, 9 (51) 1411-1422
- Martinez Palacios P., Jacquemot MP., Tapie M., Rousselet A., Diop M., Remoué C., Falque M. , Lloyd A., Jenczewski E., Lassalle G., Chévre AM., Lelandais C., Crespi M., Brabant P., Joets J., Alix K.. (2019) Assessing the Response of Small RNA Populations to Allopolyploidy Using Resynthesized Brassica napus Allotetraploids. Mol Biol Evol, 4 (36) 709-726
- Termolino P., Falque M. , Aiese Cigliano R., Cremona G., Paparo R., Ederveen A., Martin OC., Consiglio FM., Conicella C.. (2019) Recombination suppression in heterozygotes for a pericentric inversion induces the interchromosomal effect on crossovers in Arabidopsis. Plant J, 6 (100) 1163-1175
- Tourrette E., Bernardo R., Falque M. , Martin OC.. (2019) Assessing by Modeling the Consequences of Increased Recombination in Recurrent Selection of Oryza sativa and Brassica rapa. G3, 12 (9) 4169-4181
- Tourrette E., 25 novembre 2019, Unleashing genetic diversity in breeding by increasing recombination: an in silico study, PhD Thesis, Université de Paris-Diderot
- Virlouvet L., El Hage F., Griveau Y., Jacquemot MP., Gineau E., Baldy A., Legay S., Horlow C., Combes V., Bauland C., Palafre C., Falque M. , Moreau L., Coursol S., Méchin V., Reymond M.. (2019) Water Deficit-Responsive QTLs for Cell Wall Degradability and Composition in Maize at Silage Stage. Front. Plant Sci., (10) 488
2018
- Raffoux X., 2018-06-11 06/11/18, Diversité et déterminisme génétique de la recombinaison méiotique chez Saccharomyces cerevisiae, PhD thesis, Université Paris-Saclay
- Raffoux X., Bourge M., Dumas F., Martin OC., Falque M. . (2018) High-throughput measurement of recombination rates and genetic interference in Saccharomyces cerevisiae. Yeast, 6 (35) 431-442
- Raffoux X., Bourge M., Dumas F., Martin OC., Falque M. . (2018) Role of Cis, Trans, and Inbreeding Effects on Meiotic Recombination in Saccharomyces cerevisiae. Genetics, 4 (210) 1213-1226
2017
- Giraud H., Bauland C., Falque M. , Madur D., Combes V., Jamin P., Monteil C., Laborde J., Palaffre C., Gaillard A., Blanchard P., Charcosset A., Moreau L.. (2017) Linkage Analysis and Association Mapping QTL Detection Models for Hybrids Between Multiparental Populations from Two Heterotic Groups: Application to Biomass Production in Maize (Zea mays L.). G3: Genes, Genomes, Genetics, g3.300121.2017
- Giraud H., Bauland C., Falque M. , Madur D., Combes V., Jamin P., Monteil C., Laborde J., Palaffre C., Gaillard A., Blanchard P., Charcosset A., Moreau L.. (2017) Reciprocal Genetics: Identifying QTL for General and Specific Combining Abilities in Hybrids Between Multiparental Populations from Two Maize (Zea mays L.) Heterotic Groups. Genetics, 3 (207) 1167-1180
- Pelé A., Falque M. , Trotoux G., Eber F., Nègre S., Gilet M., Huteau V., Lodé M., Jousseaume T., Dechaumet S., Morice J., Poncet C., Coriton O., Martin OC., Rousseau-Gueutin M., Chèvre AM.. (2017) Amplifying recombination genome-wide and reshaping crossover landscapes in Brassicas. PLoS Genet, 5 (13)
2016
- Boutet G., Alves Carvalho S., Falque M. , Peterlongo P., Lhuillier E., Bouchez O., Lavaud C., Pilet-Nayel ML., Rivière N., Baranger A.. (2016) SNP discovery and genetic mapping using genotyping by sequencing of whole genome genomic DNA from a pea RIL population. BMC Genomics, (17)
2013
- Basu-Roy S., Gauthier F., Giraut L., Mezard C., Falque M. , Martin OC.. (2013) Hot Regions of Noninterfering Crossovers Coexist with a Nonuniformly Interfering Pathway in Arabidopsis thaliana. Genetics, 3 (195) 769-79
- Bauer E., Falque M. , Walter H., Bauland C., Camisan C., Campo L., Meyer N., Ranc N., Rincent R., Schipprack W., Altmann T., Flament P., Melchinger AE., Menz M., Moreno-Gonzalez J., Ouzunova M., Revilla P., Charcosset A., Martin OC., Schon CC.. (2013) Intraspecific variation of recombination rate in maize. Genome biology, 9 (14) R103
- Drouaud J., Khademian H., Giraut L., Zanni V., Bellalou S., Henderson IR., Falque M. , Mezard C.. (2013) Contrasted Patterns of Crossover and Non-crossover at Arabidopsis thaliana Meiotic Recombination Hotspots. PLoS genetics, 11 (9) e1003922
- Sarilar V., Martinez-Palacios P., Rousselet A., Ridel C., Falque M. , Eber F., Chevre AM., Joets J., Brabant P., Alix K.. (2013) Allopolyploidy has a moderate impact on restructuring at three contrasting transposable element insertion sites in resynthesized Brassica napus allotetraploids. The New phytologist, 2 (198) 593-604